Предлагаемый протокол включает следующие основные элементы:
1. Формулировка исследовательской гипотезы. Выполнение эксперимента для сбора наблюдаемых данных.
2. Разведочный анализ данных:
- Выявление точек-выборосов
- Проверка однородности дисперсий
- Проверка нормальности распределения данных
- Выявление избыточного количества нулевых значений
- Выявление коллинеарных переменных
- Выявление характера связи между анализируемыми переменными
- Выявление взаимодействий между переменными-предикторами
- Выявление пространственно-временных корреляций между значениями зависимой переменной
3. Применение статистического метода (модели) в соответствии с поставленной задачей и свойствами данных.
Выявление коллинеарных переменных
Для конкретизации наших дальнейших рассуждений рассмотрим набор данных sleep из пакета VIM, составленный по результатам наблюдений за процессом сна 62 млекопитающих разных видов (Allison, Chichetti, 1976). Авторы заинтересовались тем, почему потребность в сне у животных меняется от вида к виду и от каких экологических и таксономических переменных она зависит. Зависимые переменные включали продолжительность сна со сновидениями (Dream), сна без сновидений (NonD), и их сумма (sleep). Таксономические переменные включали массу тела (BodyWgt), вес мозга (BrainWgt), продолжительность жизни (Span) и время беременности (Gest).
Экологические переменные состояли из 5-бальных оценок степени хищничества животных (Pred), меры защищенности их места для сна (Exp), изменяющегося от глубокой норы до полностью открытого пространства, и показателя риска (Danger), который основывался на логической комбинации остальных двух (Pred и Exp).
sleep_imp3<-read.table("sleep_imp.dat",header=TRUE,sep="\t",dec=".")
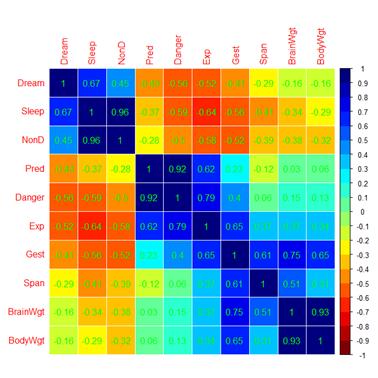
M <- cor(sleep_imp3)
Print(M)
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
BodyWgt 1.00 0.93 -0.32 -0.16 -0.29 0.31 0.65 0.06 0.34 0.13
BrainWgt 0.93 1.00 -0.38 -0.16 -0.34 0.51 0.75 0.03 0.37 0.15
NonD -0.32 -0.38 1.00 0.45 0.96 -0.39 -0.52 -0.28 -0.58 -0.50
Dream -0.16 -0.16 0.45 1.00 0.67 -0.29 -0.41 -0.43 -0.52 -0.56
Sleep -0.29 -0.34 0.96 0.67 1.00 -0.41 -0.56 -0.37 -0.64 -0.59
Span 0.31 0.51 -0.39 -0.29 -0.41 1.00 0.61 -0.12 0.37 0.06
Gest 0.65 0.75 -0.52 -0.41 -0.56 0.61 1.00 0.23 0.65 0.40
Pred 0.06 0.03 -0.28 -0.43 -0.37 -0.12 0.23 1.00 0.62 0.92
Exp 0.34 0.37 -0.58 -0.52 -0.64 0.37 0.65 0.62 1.00 0.79
Danger 0.13 0.15 -0.50 -0.56 -0.59 0.06 0.40 0.92 0.79 1.00
image(M,col=gray((32:0)/32))
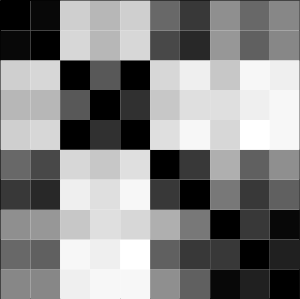
Library(corrplot)
col4 <- colorRampPalette(c("#7F0000","red","#FF7F00","yellow",
"#7FFF7F", "cyan", "#007FFF", "blue","#00007F"))
corrplot(M, method="color", col=col4(20), cl.length=21,
order = "AOE", addCoef.col="green")
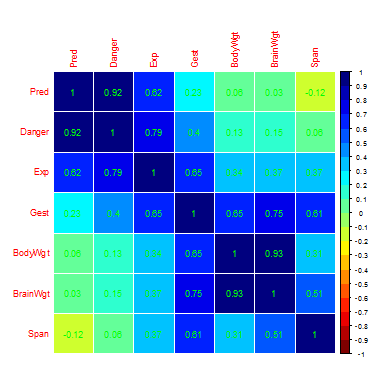
Но нас больше интересует коллинеарность независимых переменных
p.names.ind<-c("BodyWgt","BrainWgt","Span","Gest","Pred","Exp","Danger")
M1 <- cor(sleep_imp3[,p.names.ind])
corrplot(M1, method="color", col=col4(20), cl.length=21,
order = "AOE", addCoef.col="green")
Однако только проверки парной корреляции недостаточно, нужно оценить множественную – так называемый множитель уменьшения дисперсии VIF
Рассмотрим линейную модель с k независимыми переменными:
Y = β 0 + β 1 X 1 + β 2 X 2 +... + βk Xk + ε.
Стандартная ошибка определения βj может быть представлена как

где Rj 2 есть множественный R2 для регрессии Xj на остальные переменные, и s 2: оценка дисперсии ошибки
Множитель 1 / (1 − Rj 2) есть VIF (variance inflation factor), т.е. множитель увеличения дисперсии оценки. Т.е. чем сильнее зависит переменная от остальных, тем больше ошибка ее определения. В обычном случае он может быть рассчитан только для скалярных переменных.
Значение VIF более 3 считается достаточно большим для исключения переменной. Переменные исключают последовательно, начиная с переменных с максимальным значением.
m1<-lm(BodyWgt~BrainWgt+Span+Gest+Pred+Exp+Danger,data=sleep_imp3)
m1.summ<-summary(m1)
m1.VIF<-1/(1-m1.summ$adj.r.squared)
Print(round(m1.VIF,1))
[1] 11.3
m1<-lm(BodyWgt~.,data=sleep_imp3[,p.names.ind])
m1.summ<-summary(m1)
m1.VIF<-1/(1-m1.summ$adj.r.squared)
Print(round(m1.VIF,1))
[1] 11.3
Summary(m1)
Call:
lm(formula = BodyWgt ~., data = sleep_imp3[, p.names.ind])
Residuals:
Min 1Q Median 3Q Max
-1385.47 -77.01 -19.96 86.40 1078.83
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 143.67244 90.58322 1.586 0.1185
BrainWgt 1.01180 0.05732 17.651 < 2e-16 ***
Span -13.12207 2.65341 -4.945 7.52e-06 ***
Gest -0.15596 0.47445 -0.329 0.7436
Pred 43.33017 66.31123 0.653 0.5162
Exp 100.82910 45.05559 2.238 0.0293 *
Danger -124.97919 83.45893 -1.497 0.1400
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 267.4 on 55 degrees of freedom
Multiple R-squared: 0.9203, Adjusted R-squared: 0.9116
F-statistic: 105.8 on 6 and 55 DF, p-value: < 2.2e-16
all.VIF<-matrix(ncol=1,nrow=length(p.names.ind))
rownames(all.VIF)<-p.names.ind
colnames(all.VIF)<-"VIF"
for(i in 1:length(p.names.ind)) {
tmp.f<-as.formula(sprintf("%s~.",p.names.ind[i]))
m1 <-lm(tmp.f,data=sleep_imp3[,p.names.ind])
m1.summ<-summary(m1)
all.VIF[i,1]<-round(1/(1-m1.summ$adj.r.squared),1)
}
Print(all.VIF)
VIF
BodyWgt 11.3
BrainWgt 14.6
Span 2.5
Gest 3.6
Pred 7.4
Exp 4.4
Danger 11.6
cur.names<-p.names.ind
while(TRUE) {
if(length(cur.names) <= 1) break;
all.VIF<-matrix(ncol=1,nrow=length(cur.names))
rownames(all.VIF)<-cur.names
colnames(all.VIF)<-"VIF"
for(i in 1:length(cur.names)) {
tmp.f<-as.formula(sprintf("%s~.",cur.names[i]))
m1 <-lm(tmp.f,data=sleep_imp3[,cur.names])
m1.summ<-summary(m1)
all.VIF[i,1]<-round(1/(1-m1.summ$adj.r.squared),1)
}
Print(all.VIF)
if(!any(all.VIF[,1] > 3)) break
pos<-which.max(all.VIF[,1])
print(c("Exclude",cur.names[pos]))
cur.names<-cur.names[-pos]
}
VIF
BodyWgt 11.3
BrainWgt 14.6
Span 2.5
Gest 3.6
Pred 7.4
Exp 4.4
Danger 11.6
[1] "Exclude" "BrainWgt"
VIF
BodyWgt 1.7
Span 1.8
Gest 3.4
Pred 7.5
Exp 4.1
Danger 11.5
[1] "Exclude" "Danger"
VIF
BodyWgt 1.7
Span 1.8
Gest 3.4
Pred 2.0
Exp 2.8
[1] "Exclude" "Gest"
VIF
BodyWgt 1.1
Span 1.5
Pred 2.0
Exp 2.4
Library(car)
vif(lm(Sleep~BodyWgt+BrainWgt+Span+Gest+Pred+Exp+Danger,data=sleep_imp3))
BodyWgt BrainWgt Span Gest Pred Exp Danger
12.540596 16.168445 2.770471 3.990466 8.240583 4.866211 12.846658
Оценка вида связи между переменными
Data(mtcars)
| [, 1] | mpg | Miles/(US) gallon |
| [, 2] | cyl | Number of cylinders |
| [, 3] | disp | Displacement (cu.in.) |
| [, 4] | hp | Gross horsepower |
| [, 5] | drat | Rear axle ratio |
| [, 6] | wt | Weight (lb/1000) |
| [, 7] | qsec | 1/4 mile time |
| [, 8] | vs | V/S |
| [, 9] | am | Transmission (0 = automatic, 1 = manual) |
| [,10] | gear | Number of forward gears |
| [,11] | carb | Number of carburetors |
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
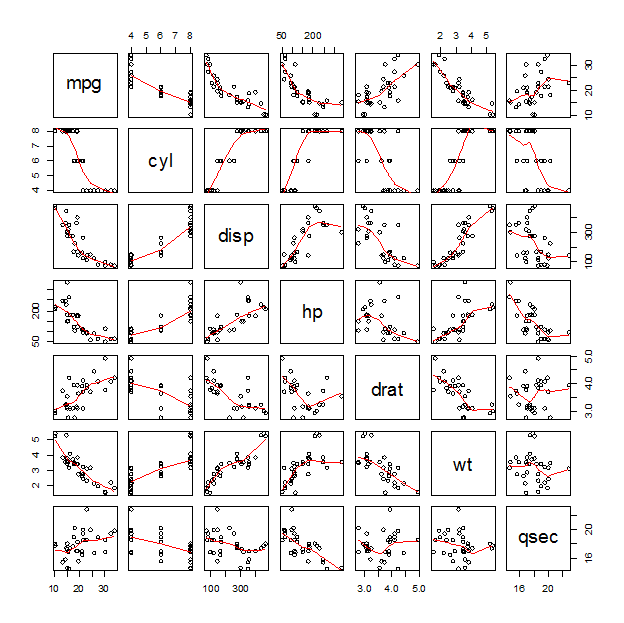
Library(lattice)
cars <- mtcars[, 1:7]
pairs(cars, panel = panel.smooth)