1 Г. А. Геращенков *, 1 Н. А. Рожнова, 1 Б. Р.Кулуев, 1 О. Ю. Кирьянова, 1 Г. Р. Гумерова, 1 А. В. Князев, 1 З. Р. Вершинина, 1 Е. В. Михайлова, 1 Д. А. Чемерис, 1 Р. Т. Матниязов, 1 Ан. Х. Баймиев, 2 И. М.Губайдуллин, 1 Ал. Х. Баймиев, 1 А. В. Чемерис
1Институт биохимии и генетики Российской академии наук,, Уфа, 450054
2Институт нефтехимии и катализа Российской академии наук, Уфа, 450075
CRISPR/Cas-технология геномного редактирования является мощным инструментом для внесения направленных изменений в ДНК различных организмов, включая растительные. Важной составляющей этой технологии является выбор мишеней в виде конкретных нуклеотидных последовательностей (протоспейсеров) в намечаемых для редактирования генах, сопряженный с дизайном гидовых РНК, осуществляемым с помощью специализированных компьютерных программ. В данном обзоре в общей сложности рассмотрено и упомянуто около ста программ дизайна гидовых РНК. Подробно рассмотрены программы дизайна гидовых РНК, позволяющие вести поиск нецелевых сайтов редактирования в геномах организмов из царства растений. Приведены также перечни web-ресурсов и off-line программных продуктов, с помощью которых можно проводить дизайн гидовых РНК разных организмов, но не позволяющих контролировать возникновение потенциальных нецелевых сайтов редактирования в геномах растений. Проведено сравнение используемых для геномного редактирования нуклеаз Cas9 и Cas12a и отмечено некоторое удобство и даже превосходство последней. Определенное внимание уделено набирающему силу редактированию отдельных азотистых оснований путем транзиций C→T и G→A без образования двуцепочечных разрывов. Указаны основные требования, предъявляемые к эффективным и высокоспецифичным гидовым РНК. Отмечены перспективные направления совершенствования программ дизайна гидовых РНК. Обсуждается также меньшая опасность нецелевого редактирования геномов растений в отличие от животных организмов. Рассмотрены особенности редактирования растительных геномов, в том числе не приводящего к созданию трансгенных растений.
|
|
Ключевые слова: CRISPR, Cas9, Cas12a, Cpf1, PAM, sgRNA, спейсер, протоспейсер, целевой сайт, нецелевой сайт, компьютерная программа, web-ресурс
*Адрес для корреспонденции: Геращенков Григорий Алексеевич. 450054. Уфа, Проспект Октября 71, ИБГ УФИЦ РАН. Электронная почта: apomixis@anrb.ru
ВВЕДЕНИЕ
CRISPR/Cas-технология геномного редактирования является мощным инструментом для внесения направленных изменений в ДНК различных организмов, включая растительные. Эта технология имеет многостадийную природу, состоящую из нескольких этапов, и каждый из них в той или иной степени определяет общий успех при достижении конечного результата. Среди этапов редактирования растительных геномов важная роль отводится способу доставки CRISPR/Cas-компонентов к месту редактирования, от чего зависит, в том числе и генетический (генно-модифицированный) статус создаваемого растения, что уже рассмотрено нами ранее [1]. Также весьма ответственным моментом является выбор мишеней в виде конкретных нуклеотидных последовательностей (протоспейсеров) внутри намечаемых для редактирования генов, сопряженный с дизайном гидовых РНК (sgRNA), осуществляемым с помощью специализированных компьютерных программ, которых написано уже значительное количество и именно они явятся предметом рассмотрения в данной работе.
|
|
Имеется целый ряд обзорных статей, посвященных программам дизайна sgRNA [2 - 9]. Нами также был подготовлен обзор на эту тему [10], причем он включал рассмотрение не только программ для дизайна sgRNA, но и баз данных по этим компонентам CRISPR/Cas-редактирования, а также программы анализа редактированных геномов. Однако часть этих обзоров подготовлена относительно давно, а с учетом очень бурно развивающейся сферы геномного редактирования с помощью CRISPR/Cas-технологии они уже не в полной мере отражают современные подходы к дизайну гидовых РНК. Так, за истекший период ряд ранних программ претерпели определенные обновления, появилось немало новых подобных ресурсов из-за чего необходимо обратить внимание на этот крайне важный этап геномного редактирования вновь. В отличие от других обзорных публикаций в данной работе будут рассмотрены практически все существующие программы дизайна sgRNA, нацеленные именно на редактирование геномов растений. Поскольку эта статья рассчитана в первую очередь на конечных пользователей таких программных продуктов, то алгоритмов, используемых разными разработчиками при написании самих программ, коснемся очень кратко. Программы анализа результатов геномного редактирования также останутся за пределами рассмотрения, поскольку должны составить тему отдельной статьи.
Что касается программ дизайна sgRNA, предназначенных для редактирования геномов иных организмов (не из царства растений), то необходимо отметить, что с помощью большинства из них дизайн sgRNA может проводиться для любых нуклеотидных последовательностей, не взирая на принадлежность к тем или иным формам Жизни. Однако такие программы не позволяют оценивать наличие в геномах конкретных растений нецелевых мест редактирования. Так, например, программный продукт CRISPR-Cas9 guide RNA design checker (https://eu.idtdna.com/site/order/designtool/index/crispr_sequence) известной фирмы Integrated DNA Technologies позволяет осуществлять дизайн sgRNA и проверку их успешности на предмет отсутствия нецелевых сайтов в геномах человека, мыши, крысы, аквариумной рыбки Danio rerio и нематоды. Для других видов можно выставить опцию «Other species», где в скобках указывается «no off-target analysis». При этом у экспериментатора в качестве некоей неполноценной альтернативы имеется возможность проверить как полное, так и неполное совпадение спейсерных последовательностей sgRNA, подобранных этой или аналогичными программами, по базе данных GenBank с использованием сервиса Nucleotide BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi). Также следует заметить, что полных геномов растений известно до сих пор все же не так много, и это в основном модельные объекты или широко возделываемые сельскохозяйственные растения, тогда как может потребоваться редактирование генома какого-либо редкого вида растения, для которого геном еще не секвенирован, даже частично. Для этих целей вполне пригодны любые программы дизайна sgRNA (перечни которых в данной статье приведены с указанием их URL-адресов), в том числе не предназначенные для выявления нецелевых мишеней в известных полных геномах растений.
|
|
Прежде чем перейти к изложению основного материала необходимо коснуться вопроса обозначения применяемых для геномного редактирования Cas-нуклеаз и их мутантных форм, созданных генно-инженерным путем, иногда сопровождаемых двух-трехбуквенными сокращениями латинских названий бактерий. В данной статье мы будем приводить те их обозначения, которые использовались самими разработчиками описываемых программ, подразумевая при этом, что под обозначением Cas9, где не оговаривается ее происхождение, но речь идет о конкретном ферменте, по умолчанию будет скрываться нуклеаза дикого типа wtSpCas9 из Streptococcus pyogenes.
CAS9/12A-НУКЛЕАЗЫ, ИСПОЛЬЗУЕМЫЕ В ГЕНОМНОМ РЕДАКТИРОВАНИИ
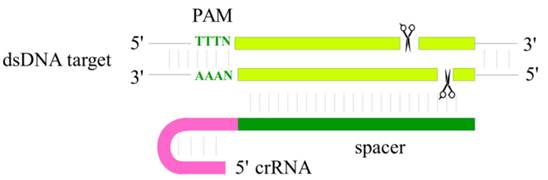
Подробное рассмотрение CRISPR/Cas-технологии геномного редактирования, включая детальный механизм действия используемых нуклеаз и различные достижения по улучшению хозяйственно-полезных признаков растений, в задачи данной статьи не входят, тем более, что этим вопросам посвящено множество обзоров [11 - 22]. Однако следует коротко остановиться на самих нуклеазах Cas9 и Cas12a (Cpf1). Основные различия между ними, как можно видеть из рис. 1, заключаются в расположении узнаваемой первой PAM-последовательности, соседствующей с протоспейсером, а также в местах разрыва цепей ДНК и структурной организацией остова гидовой РНК (crRNA и tracrRNA), образующей с тем или иным апоферментом соответствующий рибонуклеопротеидный комплекс (РНП).
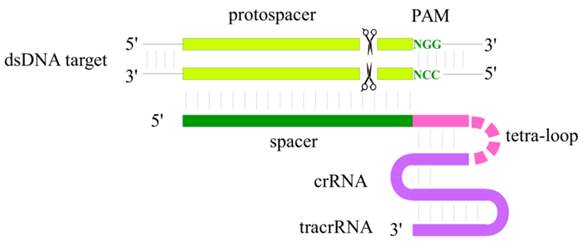
А
Б
Рис. 1. Схемы взаимодействий рибонуклеопротеидных комплексов из полноразмерных гидовых РНК с нуклеазами Cas9 (А) и Cas12a (Б) с редактируемой ДНК. (Пояснения в тексте).
Классический вариант нуклеазы Cas9 узнает PAM‑последовательность NGG‑3’, а Cpf1-нуклеаза узнает расположенную на противоположном конце протоспейсера AT‑богатую PAM‑последовательность 5’‑TTTN. Протяженность спейсерных последовательностей гидовых РНК этих нуклеаз составляют 20 и 23 нуклеотида соответственно. Наиболее важную для узнавания протоспейсера часть спейсерной последовательности, прилегающую к PAM-участку, называют «seed sequence» (в данной статье она будет упоминаться как «якорная последовательность» или «якорный участок»). Ее длина для нуклеаз из группы Cas9 составляет 8 – 12 нуклеотидов, а у Cas12a-нуклеаз она короче – всего 5 – 6 нуклеотидов. Cpf1-нуклеаза образует на некотором удалении от PAM-участка двуцепочечный разрыв с выступающими четырьмя–пятью нуклеотидами на 5’‑концах против обычно тупоконечного разрыва, осуществляемого нуклеазой Cas9 в якорном участке поблизости от PAM-последовательности. У Cpf1-нуклеазы имеется один каталитический домен RuvC, расщепляющий обе цепи ДНК, тогда как Cas9-нуклеаза для расщепления комплементарных цепей ДНК несет два каталитических домена RuvC и HNH, единичные мутации в которых (H840A в RuvC и D10A в HNH) превращают Cas9-нуклеазу в соответствующие nCas9-никазы, поскольку остающиеся неизмененными вторые домены будут разрезать только одну цепь. При одновременном нарушении обоих каталитических доменов Cas9-нуклеаза, называемая уже dCas9-нуклеазой, теряет способность к разрезанию цепей ДНК. Однако если подобную неактивную нуклеазу сшить с мономером рестрикционной эндонуклеазы Fok I, то будет образована нуклеаза FokI-dCas9, сокращенно обозначаемая также как fCas9. При использовании двух таких химерных ферментов и связывании их с редактируемой ДНК на нужном расстоянии, возникает димер Fok I, способный проявлять каталитическую активность. FokI-dCas9, наряду с nCas9 никазами, также находят применение в геномном редактировании, теоретически снижая число мест нецелевого редактирования за счет уменьшения вероятности нахождения двух довольно протяженных последовательностей разных протоспейсеров на близком расстоянии.
Еще одним типом химерных Cas-нуклеаз являются лишенные каталитической активности нуклеазы dCas или никазы nCas, несущие пришитые к ним цитидин- или аденин-деаминазы (вкупе с ними также и некоторые другие ферменты), позволяющие производить геномное редактирование отдельных азотистых оснований путем транзиций C→T и A→G без образования двуцепочечных разрывов молекул ДНК. Здесь необходимо заметить, что редактирование отдельных азотистых оснований позволяет осуществлять и нокаутное редактирование за счет формирования терминирующих кодонов (TAA, TAG, TGA) из кодонов, кодирующих глутамин (CAA и CAG), аргинин (CGA) и триптофан (TGG). Впрочем, возможно и обратное восстановление кодонов данных аминокислот из терминаторов транскрипции, что немаловажно для геномного редактирования в виде удаления ненужных мутаций.
У разных видов бактерий найдены Cas9/12a-нуклеазы, узнающие заметно отличающиеся PAM-последовательности. Немало создано подобных генетически-модифированных Cas-нуклеаз, также узнающих разные PAM-участки, что с одной стороны расширяет возможности экспериментаторов при поиске в геномах подходящих протоспейсеров, а с другой – снижает риск возникновения нецелевых сайтов редактирования при использовании нуклеаз, узнающих более протяженные PAM-последовательности, которых априори меньше в редактируемых геномах.
Серьезным отличием нуклеазы Cpf1 от Cas9-нуклеаз является то, что для ее функционирования не требуется tracrRNA и поэтому длина crRNA, достаточная для формирования активного комплекса с Cpf1-апоферментом, ограничивается всего 42 - 44 нуклеотидами против обычно используемой для Cas9-нуклеазы полноразмерной гидовой РНК длиной около 100 нуклеотидов, состоящей из crRNA, tracrRNA и искусственной тетрануклеотидной GAAA‑петли. Благодаря укороченному размеру остова гидовой РНК у нуклеазы Cpf1 этот компонент рибонуклеопротеидного комплекса удобнее синтезировать химическим способом [23], хотя подобный подход используется и для нуклеазы Cas9 [24], что заметно упрощает для экспериментатора задачи геномного редактирования, тем более, что готовые ферменты можно приобрести уже у ряда фирм. Обнаружено, что химически синтезированная гидовая РНК лучше синтезированной энзиматически, поскольку последняя при геномном редактировании с помощью рибонуклеопротеидных комплексов с Cpf1-нуклеазой приводила к плохообъяснимым инсерциям фрагментов ДНК [25]. Показано также, что Cas12a-нуклеаза характеризуется повышенной специфичностью действия [26], что делает ее использование предпочтительным, хотя пока по масштабу применения она заметно уступает различным Cas9-нуклеазам. Можно предположить, что более высокая специфичность Cas12a-нуклеазы связана с тем, что она расщепляет ДНК относительно далеко от PAM-участка и происходящие «плавление» цепей ДНК, а также продвижение так называемой R-петли, чувствительные к возможным неспариваниям и идущие постепенно, могут в какой-то момент из-за наличия некомплементарных нуклеотидов прекратиться, и рибонуклеотидный комплекс абортирует с неполностью совпадающего нецелевого сайта, тогда как в случае Cas9-нуклеазы места расщепления обеих цепей ДНК находятся вблизи от PAM-участка и теоретически могут быть расщеплены, не дожидаясь формирования полноценной R-петли в нецелевом сайте редактирования.
ОСНОВНЫЕ ТРЕБОВАНИЯ, ПРЕДЪЯВЛЯЕМЫЕ К ДИЗАЙНУ ГИДОВЫХ РНК
Для успешного геномного редактирования и исключения нецелевых мутаций при эффективном произведении целевых требуется выполнить качественный дизайн sgRNA. Для этого необходим анализ всего генома конкретного организма (в тех случаях, когда его последовательность известна) на предмет наличия в нем сходных с редактируемым участком нуклеотидных последовательностей. Поскольку неспаривание отдельных нуклеотидов и даже наличие небольших инделов (как в ДНК, так и в РНК-цепях) теоретически может не послужить препятствием для произведения такого нецелевого редактирования, то во внимание следует принимать не только 100%-ное совпадение нуклеотидных последовательностей sgRNA с потенциальными протоспейсерами. Таким образом, выбранные гидовые РНК не должны иметь полностью совпадающих нецелевых сайтов в редактируемом геноме, а наличие гомологичных лишь частично, должно быть сведено к минимуму. Причем особое внимание нужно уделять якорной части, где замены нуклеотидов наиболее критичны. Следует также обращать внимание на локализацию потенциальных нецелевых сайтов – в кодирующих областях, в интронах, промоторах, межгенных участках – от этого зависит серьезность исходящей опасности такого редактирования. Для более эффективного нокаутирования конкретных генов предпочтительнее выбирать расположение редактируемого участка в первом экзоне (если таковые имеются) и на расстоянии 150 – 500 пар нуклеотидов от стартового кодона. Для небольших белков данный диапазон желательно должен составлять от 100 до 300 пар нуклеотидов. Стремясь исключить появление усеченного по C-концу, но остающегося функциональным белка, и располагая место редактирования ближе к NH2-концу, нельзя забывать о возможном лидерном пептиде, нарушения в котором не затронут сам белок, но изменят его компартментализацию.
Еще в 2013 г. анализ более 700 sgRNA позволил выработать ряд рекомендаций по их подбору, снижающему возможность нецелевого редактирования и написать одну из первых программ для дизайна sgRNA [27]. Главным требованием было исключение нахождения рядом с нецелевым участком, имеющим гомологию с целевым, PAM-последовательности, поскольку именно на нее первоначально ориентируется нуклеаза. Далее уже для мест с PAM-последовательностью рекомендовалось исключить полную гомологию нецелевого сайта с целевым и допускать наличие в нем не менее трех замен нуклеотидов, при этом, по крайней мере, две из них должны находиться в области, прилегающей к PAM-последовательности. К тому же желательно, чтобы такими неспариваниями характеризовались соседние нуклеотиды или они отстояли бы друг от друга не более чем на четыре положения. В дальнейшем анализ 1841 варианта sgRNA продемонстрировал предпочтительное нахождение тех или иных азотистых оснований в спейсерной последовательности и дал возможность создать предиктивную модель их активности и выработать определенные правила [28], названные позже как Rule Set 1. Так, было обнаружено, что Cas9-нуклеаза предпочитает, чтобы вариабельный нуклеотид PAM-последовательности был цитозином, но он крайне нежелателен в качестве соседствующего с ней уже в участке ДНК, взаимодействующим с sgRNA, где предпочитетелен гуанин. В своей следующей работе [29] эти авторы продолжили изучение целевого и нецелевого редактирования sgRNA и разработали усовершенствованные правила Rule Set 2, послужившие основой для целого ряда программ дизайна sgRNA. В дальнейшем при усовершенствовании программных продуктов дизайна sgRNA и создании новых стли активно применяться алгоритмы, основанные на технологиях глубокого машинного обучения, позволяющие вести улучшенный поиск уникальных протоспейсеров с уменьшенной вероятностью возникновения нецелевых сайтов редактирования [30 - 36].
Как будет видно из дальнейшего изложения, программы дизайна sgRNA, предназначенные для редактирования геномов растительных организмов, заметно отличаются друг от друга, однако ряд моментов носят общий характер. К таковым относятся протяженности спейсерных областей sgRNA, хотя и варьирующие при дизайне в пределах от 15 до 25 нуклеотидов, но характеризующиеся предустановленными во многих программах их длинами в 20 и 23 нуклеотида (по умолчанию) для нуклеаз Cas9 и Cas12a (Cpf1) соответственно. Считается, что использование sgRNA с укороченным спейсером несколько снижает неспецифичное редактирование, однако чрезмерное укорочение может обернуться потерей каталитической активности Cas-нуклеаз вообще. Немаловажное значение имеет и нуклеотидный состав протоспейсеров. Не рекомендуется выбирать подобные участки как с высоким, так и с низким GC-содержанием, желательно укладываясь в границы от 40 до 60%, хотя и 20-80% считаются приемлемыми. Необходимо исключать гомополимерные участки из 4 и более одинаковых нуклеотидов, а наличие подряд четырех тиминов в протоспейсере (или даже трех) должно исключаться в случае наработки sgRNA с помощью транскрипции, поскольку эта последовательность способна выполнить роль терминатора данного процесса. Также необходимо учитывать системы экспрессии, применяемые для наработки sgRNA и требующие для большей эффективности транскрипции наличия на 5’-конце двух гуанинов в случае использования РНК-полимеразы под контролем Т7 промотора или одного гуанина для промотора U6. Нуклеотидная последовательность выбранного участка должна исключать возникновение неправильной пространственной структуры всей молекулы sgRNA. При подборе протоспейсеров в месте двухцепочечного разрыва и последующей репарации желательно предусматривать нахождение сайтов рестрикционных эндонуклеаз, которые при расщеплении ампликонов, образованных с помощью ПЦР с фланкирующими праймерами, служат контролем произошедшего геномного редактирования, предшествуя секвенированию данного участка генома. При проведении экспериментов по CRISPR/Cas-редактированию в нокин вариантах подобраннные протоспейсеры, в том числе с учетом небольшого количества замен, должны отсутствовать в донорной ДНК.
Недавно анализ большого числа неуспешных гидовых РНК показал, что большинство из них содержат в проксимальной части спейсерных последовательностей среди четырех, непосредственно прилегающих к PAM-участку нуклеотидов, мотивы TT и GCC, снижающие эффективность редактирования на порядок, что позволяет задуматься об исключении таковых при дизайне sgRNA [37]. Еще ранее разработчики программы дизайна гидовых РНК WU-CRISPR [38], сообщали, что если четыре нуклеотида спейсерной последовательности, прилегающие к PAM-участку, обогащены пиримидинами, то эффективность геномного редактирования с участием таковых sgRNA резко снижается по причине того, что в этом случае они могут спариваться с определенными нуклеотидами остова гидовой РНК, удлиняя тем самым одну из ее петлевых структур и выключая, соответственно, важную часть якорной последовательности из взаимодействия с сайтом редактирования.
НУКОТОРЫЕ ОСОБЕННОСТИ РЕДАКТИРОВАНИЯ ГЕНОМОВ ОРГАНИЗМОВ ИЗ ЦАРСТВА РАСТЕНИЙ
При редактировании (в нокаутном варианте) растительных геномов появление помимо целевых мутаций еще иных мест, подвергнувшихся редактированию не так катастрофично, как для животных организмов. Причиной тому служат крупные размеры геномов у многих растений, содержащие значительную долю некодирующих участков, их зачастую полиплоидный характер, включая и так называемые палеополиплоиды (ныне считающиеся диплоидными формами), увеличенная копийность многих генов, нередко встречающееся эндополиплоидное состояние отдельных клеток и тканей, вызванное эндоредупликацией. Другой причиной меньших опасений нецелевого редактирования геномов растений является возможность для большинства видов относительно легкой сегрегации нежелательных признаков в ряду поколений, а также проведение возвратных и насыщающих скрещиваний. Наконец, существует большой опыт случайного мутагенеза под воздействием радиации или химических агентов, в результате которого происходит огромное количество мутаций в непредсказуемых местах генома, а отдельные растения при этом не только выживают, но, случается, приобретают и полезные признаки.
Фактически нецелевое геномное CRISPR/Cas-редактирование (не являющееся все же полностью случайным) можно считать, с определенными допущениями, мало отличающимся от химического или радиационного мутагенеза, после которого получившиеся новые линии и сорта растений за ГМО не признаются. Теоретически можно представить внесение многочисленных мутаций в различные места генома растений, например, с помощью неких вырожденных sgRNA (аналогично вырожденным праймерам, отжигающим на множестве мест [39], способными произвести редактирование такого же множества протоспейсеров в ДНК того или иного вида растений, что позволит получить среди новых форм (те, что выживут) даже характеризующиеся улучшенными полезными признаками. Возможно вместо обычных Cas-нуклеаз, приводящих к образованию инделов, для такой цели более удобным окажется относительно «мягкое» редактирование отдельных азотистых оснований с помощью деаминаз, сшитых с неактивными Cas-нуклеазами. И тогда такие растения теоретически также не должны считаться генетически-модифицированными в виду нецелевого воздействия аналогично мутагенезированным химическими агентами или радиацией. Однако трудоемкость, длительность, да и стоимость такого подхода для множественного и практически случайного редактирования значительно превышает обработку семян испытанными мутагенами и вряд ли когда он будет использоваться для создания новых мутантных и при этом улучшенных форм растений.
Весьма показательно название одной из недавно опубликованных статей “CRISPR/Cas precision: do we need to worry about off-targeting in plants?”, в котором авторы прямо задаются вопросом – а надо ли вообще беспокоиться о нецелевых местах CRISPR/Cas редактирования в геномах растений? [40]. При этом они ссылаются на крупномасштабное исследование других авторов, не обнаруживших массовых мутаций после геномного редактирования. Так было проведено полногеномное секвенирование 49 растений риса, из которых - 34 растения были редактированы нуклеазой Cas9 и 15 растений, для редактирования которых использовалась нуклеаза Cpf1, наряду с еще 20 контрольными растениями [41]. В результате было обнаружено, что практически все мутации (от 102 до 148 однонуклеотидных замен и от 32 до 83 инделов на растение) возникли естественным путем, и только одна мутация оказалась следствием нецелевого редактирования Cas9 нуклеазой, а для этого фермента ими использовалось 12 sgRNA и 3 sgRNA для Cpf1 нуклеазы, нецелевого редактирования для которой вовсе не обнаружено. Это свидетельствует, наверное, в первую очередь, о хорошо подобранных этими авторами sgRNA, а также о том, что нецелевое редактирование растительных геномов происходит реже, чем обычные мутации, случающиеся как показано, например, для арабидопсиса, преимущественно в виде транзиций и небольших инделов в среднем с частотой около 10-9 на сайт за одно поколение [42]. В одной из статей [43] была приведена подборка данных по нескольким видам растений, свидетельствующая, что в большинстве случаев редактирование с помощью CRISPR/Cas систем, как работающих транзиентно, так и после их внедрения в геномы растений, было целевым. Ранее отмечалось, что нецелевое редактирование имеет на самом деле меньший масштаб, чем предполагалось, причем это наблюдение касалось разных организмов [38].
Здесь необходимо заметить, что эффективность (полнота) геномного редактирования и его специфичность (воздействие преимущественно или исключительно на целевые сайты) фактически противоборствуют друг с другом. Так, эффективность редактирования намеченных мест в геноме будет тем выше, чем дольше будет в клетке работать РНП на основе той или иной Cas-нуклеазы и гидовой РНК, одновременно увеличивая вероятность нецелевого редактирования. А продолжительность работы РНП зависит от выбранной процедуры доставки, которая для растений может как обеспечить встраивание в геном всех или только части CRISPR/Cas-компонентов (постоянное воздействие), либо привести к их транзиентной экспрессии (пролонгированное воздействие), так и произвести только кратковременное воздействие, когда доставляется непросредственно готовый рибонуклеопротеидный комплекс. В последнем случае имеет место так называемая бесплазмидная доставка, дополнительным преимуществом которой, помимо технической быстроты процедуры редактирования, является невозможность встраивания чужеродной ДНК (при нокаутном редактировании) в геном растения, которое принципиально не может стать в этом случае трансгенным. И подобные подходы в последние годы получают свое развитие [44-47]. Причем такой способ бесплазмидного редактирования безусловно накладывает отпечаток на дизайн sgRNA в плане меньшего опасения возникновения нецелевых мутаций.
С учетом увеличенной копийности отдельных генов, вариаций нуклеотидных последовательностей, а также полиплоидной природы у многих видов растений для более эффективного нокаутного редактирования следует подбирать по несколько sgRNA на каждый ген в расчете, что, по крайней мере, в разных копиях того или иного гена смогут произвести редактирование ни одна, так другая гидовая РНК.
КОМПЬЮТЕРНЫЕ ПРОГРАММЫИ WEB-РЕСУРСЫДЛЯ ДИЗАЙНА sgRNA
При осуществлении дизайна sgRNA важным моментом является прогнозирование наличия нецелевых сайтов при CRISPR/Cas-редактировании геномов. Для этого программы дизайна sgRNA должны иметь доступ к соответствующим геномным последовательностям. В таблице 1 представлена краткая информация о некоторых программных продуктах для дизайна sgRNA, содержащая также сведения о количестве геномов, в том числе из царства растений, используемых для анализа. При этом для отдельных программ по просьбе пользователей разработчики готовы включить дополнительные геномы.
Таблица 1
Программы дизайна sgRNA, сопровождаемые анализом потенциальных нецелевых сайтов для таковых в геномах растений
| Программный продукт (по алфавиту) | URL адрес | кол-во * геномов из царства растений | кол-во * прочих геномов | Ссылка |
| Benchling | https://benchling.com/ | |||
| Breaking Cas | https://bioinfogp.cnb.csic.es/tools/breakingcas/ | Ensembl ** | Ensembl | |
| CCTop | https://crispr.cos.uni-heidelberg.de | |||
| CGAT | https://cbc.gdcb.iastate.edu/cgat/ | |||
| CHOPCHOP | https://chopchop.cbu.uib.no | 18 *** | 144 *** | 51, 52 |
| CLD | https://github.com/boutroslab/cld | Ensembl ** | Ensembl | |
| CRISPOR | https://crispor.tefor.net | 94 *** | 181 *** | 54, 55 |
| CRISPRdirect | https://crispr.dbcls.jp | |||
| CRISPR-GE**** | https://skl.scau.edu.cn | |||
| crispr grna design tool | https://www.atum.bio/eCommerce/cas9/input?multipleContacts=false | |||
| CRISPR MultiTargeter | https://www.multicrispr.net | 58, 59 | ||
| CRISPR-P CRISPR-Local | https://cbi.hzau.edu.cn/CRISPR2/ https://crispr.hzau.edu.cn/CRISPR-Local/ | 60 - 62 | ||
| CRISPR-PLANT | https://www.genome.arizona.edu/crispr/ | 35, 63 | ||
| CRISPR Primer Designer | https://plantsignal.cn/CRISPR/crispr_primer_designer.html | |||
| CRISPR RGEN Tools | https://www.rgenome.net | 106 *** | 185 *** | 65 – 70 |
| CRISTA | https://crista.tau.ac.il | |||
| CT-Finder CRISPR-DT CRISPR-RT | https://bioinfolab.miamioh.edu/ct-finder/ https://bioinfolab.miamioh.edu/CRISPR-DT/ https://bioinfolab.miamioh.edu/CRISPR-RT/ | 36, 71, 72 | ||
| DESKGEN | https://www.deskgen.com/landing/cloud | |||
| Dharmacon CRISPR Design Tool | https://dharmacon.horizondiscovery.com/gene-editing/crispr-cas9/crispr-design-tool/ | |||
| E-CRISP | https://www.e-crisp.org/E-CRISP/ | |||
| GT-Scan | https://gt-scan.csiro.au | 11 *** | 41 *** | |
| PhytoCRISP-Ex | https://www.phytocrispex.biologie.ens.fr/CRISP-Ex/ | |||
| Synthego CRISPR Design Tool | https://design.synthego.com/#/ | Ensembl ** | Ensembl | |
| WheatCrispr | https://crispr.bioinfo.nrc.ca/WheatCrispr/ |
* - для многих программ периодически производится их обновление и при этом, как правило, увеличивается список доступных для анализа геномов, поэтому к моменту выхода данной статьи указанные здесь числа, актуальные на начало апреля 2019 г., могут служить лишь приблизительным ориентиром.
** - количество доступных геномов соответствует таковым, находящимся в различных разделах базы данных Ensembl, например в https://plants.ensembl.org/index.html для растений.
*** - есть возможность добавления дополнительного генома, связавшись с разработчиками.
**** - не надо путать с базой данных CrisprGE (https://crdd.osdd.net/servers/crisprge/) и программой ge-CRISPR (https://bioinfo.imtech.res.in/manojk/gecrispr/index.php)
В таблице 2 суммированы основные характеристики (более 30) описанных ниже программ дизайна sgRNA, сгруппированные в блоки (оттененные разным фоном), касающиеся Cas-нуклеаз, протоспейсеров, вариантов ввода предназначенных для редактирования последовательностей ДНК/РНК, а также особенностей выдаваемых результатов. Помимо этой информации в данной таблице приводятся сведения о возможной работе некоторых программ в автономном режиме и возможности валидации ранее подобранных sgRNA, в том числе иными программами дизайна. Некоторые программы обеспечивают еще больше вариаций анализа подобранных sgRNA, чем отражено в данной таблице, но о них будет говориться при более детальном описании этих программ.
Общий порядок дизайна sgRNA для большинства программ в целом схож. Для тех программных продуктов (для которых возможны определенные опции) поиск протоспейсеров заключается в выборе конкретных Cas-нуклеаз с указанием особенностей спейсерной области sgRNA (длины, неспариваний, инделов, GC-состава и пр.). При этом потенциальные протоспейсеры выявляются в намечаемой для редактирования последовательности генома, которая берется в анализ разными способами: ввод нуклеотидной последовательности через буфер обмена в FASTA-формате или в текстовом формате; загрузка файла; указание конкретных генов с помощью идентификатора (наименование гена, Accession number), а также с использованием геномных координат. Ряд программ допускает ввод множественных последовательностей для дизайна sgRNA, пригодных для редактирования одинаковых консервативных участков геномов разных (эволюционно близких) видов. После завершения ввода редактируемой последовательности, выбора Cas-нуклеаз и прочих параметров, включая опциональные, запускается поиск, по завершению которого программы выдают все потенциальные протоспейсеры, ранжированные или неранжированные по их пригодности, и другую информацию.
Таблица 2.
Основные характеристики некоторых программ дизайна sgRNA для CRISPR/Cas-редактирования геномов организмов из царства растений