Мастицкий, глава 7
РЕГРЕССИОННЫЕ МОДЕЛИ ЗАВИСИМОСТЕЙ МЕЖДУ КОЛИЧЕСТВЕННЫМИ ПЕРЕМЕННЫМИ
Простая линейная регрессия
Модель множественной регрессии и выбор ее спецификации
Набор данных RIKZ, модель зависимости видового обилия от NAP и beach
Описание набора данных RIKZ
| Sample | Номер образца |
| C1 | Chaetognatha species Щетинкочелюстны́е, или морски́е стре́лки |
| P1-P25 | Polychaeta species Многощетинковые черви, или полихеты |
| N1 | Nemertini species лентовидные черви |
| CR1-CR28 | Crustacea species Ракообразные |
| M1-M17 | Mollusca species Моллюски |
| I1-I5 | Insecta species |
| week | Неделя Июня (1-4), когда собирались данные |
| angle1 | Угол наклона станции |
| angle2 | Угол наклона для всего пляжа |
| exposure | Индекс, отражающий воздействие волн, длину береговой линии, уклон, мехсостав, и глубину анаэробного слоя (для пляжа) |
| salinity | Средняя соленость (для пляжа) |
| temperature | Температура (для пляжа) |
| NAP | Высота станции наблюдения по отношению к среднему уровню прилива |
| penetrability | Проницаемость |
| grainsize | Размер зерен песка |
| humus | Гумус (органика) |
| chalk | Мел |
| sorting1 | |
| Beach | Пляж |
p.lm0<-lm(lAbundance~1,data=p1)
Summary(p.lm0)
Call:
lm(formula = lAbundance ~ 1, data = p1)
Residuals:
Min 1Q Median 3Q Max
-3.3132 -1.1159 0.3504 0.9495 3.5380
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.3132 0.2465 13.44 <2e-16 ***
Residual standard error: 1.654 on 44 degrees of freedom
p.lm1<-lm(lAbundance~NAP,data=p1)
Summary(p.lm1)
Call:
lm(formula = lAbundance ~ NAP, data = p1)
Residuals:
Min 1Q Median 3Q Max
-2.1857 -0.9034 -0.3724 0.6551 4.2803
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.6196 0.2243 16.136 < 2e-16 ***
NAP -0.8813 0.2151 -4.097 0.000182 ***
Residual standard error: 1.419 on 43 degrees of freedom
Multiple R-squared: 0.2808, Adjusted R-squared: 0.264
F-statistic: 16.79 on 1 and 43 DF, p-value: 0.0001816
p.lm2<-lm(lAbundance~fBeach,data=p1)
Summary(p.lm2)
Call:
lm(formula = lAbundance ~ fBeach, data = p1)
Residuals:
Min 1Q Median 3Q Max
-2.8675 -0.7139 0.1667 0.8797 3.0731
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 4.5441 0.6713 6.769 6.6e-08 ***
fBeach2 0.8261 0.9493 0.870 0.3899
fBeach3 -1.7023 0.9493 -1.793 0.0814.
fBeach4 -1.5526 0.9493 -1.635 0.1107
fBeach5 -1.9656 0.9493 -2.071 0.0456 *
fBeach6 -1.1555 0.9493 -1.217 0.2315
fBeach7 -1.7507 0.9493 -1.844 0.0734.
fBeach8 -1.6766 0.9493 -1.766 0.0859.
fBeach9 -2.1013 0.9493 -2.213 0.0333 *
Residual standard error: 1.501 on 36 degrees of freedom
Multiple R-squared: 0.3259, Adjusted R-squared: 0.1761
F-statistic: 2.176 on 8 and 36 DF, p-value: 0.05324
p.lm3<-lm(lAbundance~NAP+fBeach,data=p1)
Summary(p.lm3)
Call:
lm(formula = lAbundance ~ NAP + fBeach, data = p1)
Residuals:
Min 1Q Median 3Q Max
-2.7491 -0.7264 -0.1494 0.7705 2.6755
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 4.1657 0.5842 7.130 2.6e-08 ***
NAP -0.7900 0.2112 -3.741 0.000656 ***
fBeach2 1.4213 0.8292 1.714 0.095351.
fBeach3 -1.3235 0.8200 -1.614 0.115517
fBeach4 -0.7391 0.8423 -0.877 0.386249
fBeach5 -1.1094 0.8454 -1.312 0.197942
fBeach6 -0.5924 0.8276 -0.716 0.478870
fBeach7 -0.6848 0.8622 -0.794 0.432384
fBeach8 -0.9011 0.8398 -1.073 0.290589
fBeach9 -1.2716 0.8435 -1.508 0.140619
Residual standard error: 1.287 on 35 degrees of freedom
Multiple R-squared: 0.5185, Adjusted R-squared: 0.3946
F-statistic: 4.187 on 9 and 35 DF, p-value: 0.0009843
p.lm4<-lm(lAbundance~NAP*fBeach,data=p1)
Summary(p.lm4)
Call:
lm(formula = lAbundance ~ NAP * fBeach, data = p1)
Residuals:
Min 1Q Median 3Q Max
-1.60286 -0.55017 0.08262 0.47421 1.65095
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 4.8577 0.4738 10.252 8.3e-11 ***
NAP 0.6547 0.5502 1.190 0.244457
fBeach2 0.2477 0.6281 0.394 0.696440
fBeach3 -2.0153 0.6161 -3.271 0.002925 **
fBeach4 -1.1946 0.6585 -1.814 0.080798.
fBeach5 -1.2550 0.6920 -1.814 0.080879.
fBeach6 -1.4717 0.6213 -2.369 0.025265 *
fBeach7 0.3950 0.8030 0.492 0.626792
fBeach8 -1.3893 0.6386 -2.175 0.038523 *
fBeach9 -1.6820 0.6617 -2.542 0.017077 *
NAP:fBeach2 0.3105 0.7082 0.438 0.664617
NAP:fBeach3 -1.9778 0.7247 -2.729 0.011037 *
NAP:fBeach4 -1.8742 0.6936 -2.702 0.011761 *
NAP:fBeach5 -2.3482 0.7579 -3.098 0.004511 **
NAP:fBeach6 -0.6434 0.6496 -0.991 0.330698
NAP:fBeach7 -3.4809 0.8082 -4.307 0.000196 ***
NAP:fBeach8 -1.8503 0.6440 -2.873 0.007826 **
NAP:fBeach9 -1.9379 0.6939 -2.793 0.009486 **
Residual standard error: 0.8804 on 27 degrees of freedom
Multiple R-squared: 0.8261, Adjusted R-squared: 0.7166
F-statistic: 7.543 on 17 and 27 DF, p-value: 2.341e-06
p.lm5<-lm(lAbundance~fBeach/NAP,data=p1)
Summary(p.lm5)
Call:
lm(formula = lAbundance ~ fBeach/NAP, data = p1)
Residuals:
Min 1Q Median 3Q Max
-1.60286 -0.55017 0.08262 0.47421 1.65095
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 4.85773 0.47381 10.252 8.30e-11 ***
fBeach2 0.24767 0.62810 0.394 0.69644
fBeach3 -2.01533 0.61606 -3.271 0.00293 **
fBeach4 -1.19456 0.65850 -1.814 0.08080.
fBeach5 -1.25503 0.69203 -1.814 0.08088.
fBeach6 -1.47174 0.62133 -2.369 0.02526 *
fBeach7 0.39499 0.80305 0.492 0.62679
fBeach8 -1.38928 0.63862 -2.175 0.03852 *
fBeach9 -1.68196 0.66170 -2.542 0.01708 *
fBeach1:NAP 0.65471 0.55024 1.190 0.24446
fBeach2:NAP 0.96517 0.44591 2.164 0.03944 *
fBeach3:NAP -1.32305 0.47161 -2.805 0.00921 **
fBeach4:NAP -1.21946 0.42224 -2.888 0.00755 **
fBeach5:NAP -1.69345 0.52123 -3.249 0.00309 **
fBeach6:NAP 0.01126 0.34525 0.033 0.97421
fBeach7:NAP -2.82617 0.59196 -4.774 5.59e-05 ***
fBeach8:NAP -1.19562 0.33471 -3.572 0.00136 **
fBeach9:NAP -1.28320 0.42275 -3.035 0.00527 **
Residual standard error: 0.8804 on 27 degrees of freedom
Multiple R-squared: 0.8261, Adjusted R-squared: 0.7166
F-statistic: 7.543 on 17 and 27 DF, p-value: 2.341e-06
p.lm6<-lm(lAbundance~fBeach:NAP+NAP,data=p1)
Summary(p.lm6)
Call:
lm(formula = lAbundance ~ fBeach:NAP + NAP, data = p1)
Residuals:
Min 1Q Median 3Q Max
-1.98065 -0.79573 0.04075 0.54003 2.70131
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.82131 0.19134 19.971 <2e-16 ***
NAP -0.01480 0.59473 -0.025 0.9803
fBeach2:NAP 1.39208 0.81618 1.706 0.0969.
fBeach3:NAP -1.30882 0.84483 -1.549 0.1303
fBeach4:NAP -1.27892 0.77326 -1.654 0.1071
fBeach5:NAP -1.81984 0.81700 -2.227 0.0324 *
fBeach6:NAP -0.04903 0.74034 -0.066 0.9476
fBeach7:NAP -1.77308 0.78514 -2.258 0.0303 *
fBeach8:NAP -1.28919 0.72458 -1.779 0.0839.
fBeach9:NAP -1.57728 0.77131 -2.045 0.0484 *
Residual standard error: 1.12 on 35 degrees of freedom
Multiple R-squared: 0.6351, Adjusted R-squared: 0.5412
F-statistic: 6.767 on 9 and 35 DF, p-value: 1.459e-05
Критерии выбора моделей оптимальной сложности
Информационный критерий Акаике

AIC(p.lm0,p.lm1,p.lm2,p.lm3,p.lm4,p.lm5,p.lm6)
df AIC
p.lm0 2 175.9660
p.lm1 3 163.1350
p.lm2 10 174.2165
p.lm3 11 161.0818
p.lm4 19 131.2571
p.lm5 19 131.2571
p.lm6 11 148.6059
Сравнение вложенных моделей
Асимптотические оценки значимости могут быть неверными, особенно на больших выборках. Поэтому можно использовать процедуру сравнения вложенных моделей. Обратите внимание, что модель p.lm2 отсутствует в списке, поскольку не вложена в p.lm1.
Anova(p.lm0,p.lm1,p.lm3,p.lm4)
Analysis of Variance Table
Model 1: lAbundance ~ 1
Model 2: lAbundance ~ NAP
Model 3: lAbundance ~ NAP + fBeach
Model 4: lAbundance ~ NAP * fBeach
Res.Df RSS Df Sum of Sq F Pr(>F)
1 44 120.332
2 43 86.546 1 33.786 43.5850 4.404e-07 ***
3 35 57.945 8 28.601 4.6120 0.0012356 **
4 27 20.930 8 37.015 5.9689 0.0001942 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Последовательное удаление переменных с оценкой значимости
drop1(p.lm4,test="F")
Single term deletions
Model:
lAbundance ~ NAP * fBeach
Df Sum of Sq RSS AIC F value Pr(>F)
<none> 20.930 1.5527
NAP:fBeach 8 37.015 57.945 31.3773 5.9689 0.0001942 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
drop1(p.lm3,test="F")
Single term deletions
Model:
lAbundance ~ NAP + fBeach
Df Sum of Sq RSS AIC F value Pr(>F)
<none> 57.945 31.377
NAP 1 23.166 81.111 44.512 13.9929 0.0006563 ***
fBeach 8 28.601 86.546 33.431 2.1595 0.0557695.
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Напомним, что drop1() работает правильнее, чем anova():
anova(p.lm3,test="F")
Analysis of Variance Table
Response: lAbundance
Df Sum Sq Mean Sq F value Pr(>F)
NAP 1 33.786 33.786 20.4074 6.826e-05 ***
fBeach 8 28.601 3.575 2.1595 0.05577.
Residuals 35 57.945 1.656
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
p.lm3a<-lm(lAbundance~fBeach+NAP,data=p1)
anova(p.lm3a,test="F")
Analysis of Variance Table
Response: lAbundance
Df Sum Sq Mean Sq F value Pr(>F)
fBeach 8 39.221 4.9026 2.9613 0.0122128 *
NAP 1 23.166 23.1661 13.9929 0.0006563 ***
Residuals 35 57.945 1.6556
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Процедуры диагностики моделей множественной регрессии
Для диагностики нужно проверить гипотезы, использованные для построения модели. Используем для примера модель p.lm3.
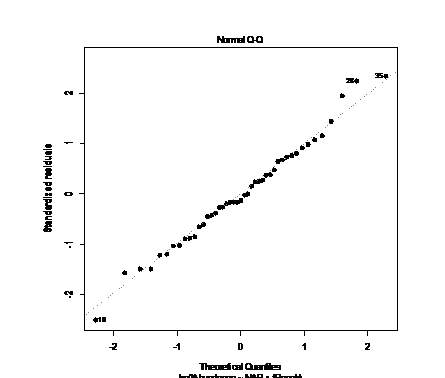
1) Нормальность остатков
# остатки против модели
plot(p.lm3,pch=20,lwd=3,cex=1.5,which=1)
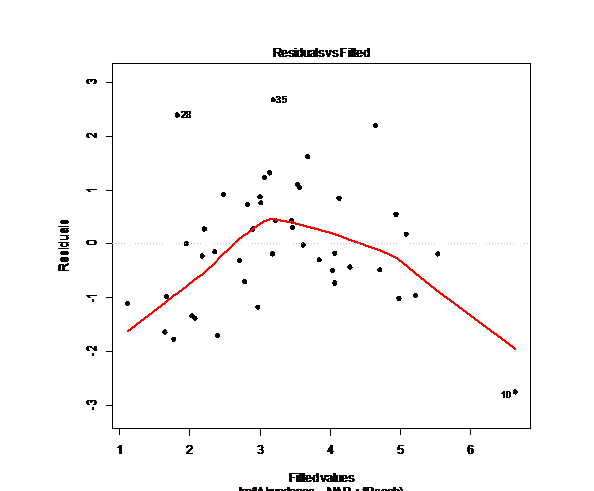
# распределение остатков в сравнении с нормальным
plot(p.lm3,pch=20,lwd=3,cex=1.5,which=2)
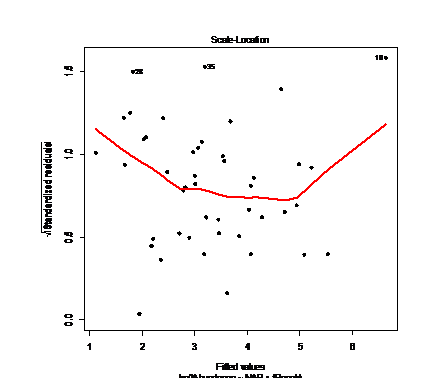
# равномерность дисперсии остатков
plot(p.lm3,pch=20,lwd=3,cex=1.5,which=3)
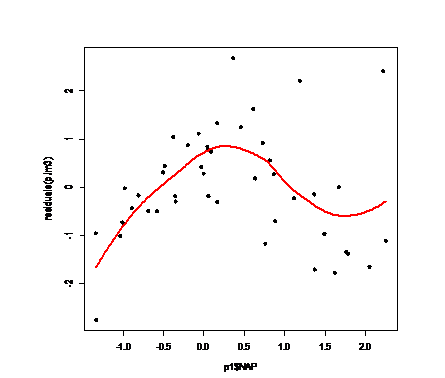
#отсуствие остатков завивисмости от переменных
# (в том числе и не включенных в модель)
plot(p1$NAP,residuals(p.lm3),pch=20,cex=1.5)
tmp.m<-loess(residuals(p.lm3)~NAP,data=p1)
tmp.x<-data.frame(NAP=seq(min(p1$NAP),max(p1$NAP),len=200))
tmp.y<-predict(tmp.m,newdata=tmp.x)
lines(tmp.x$NAP,tmp.y,lwd=3,col="red")
plot(p$salinity,residuals(p.lm3),pch=20,cex=1.5)
tmp.m<-loess(residuals(p.lm3)~salinity,data=p)
tmp.x<-data.frame(salinity=seq(min(p$salinity),max(p$salinity),len=200))
tmp.y<-predict(tmp.m,newdata=tmp.x)
lines(tmp.x$salinity,tmp.y,lwd=3,col="red")
# равномерность дисперсии остатков
boxplot(residuals(p.lm3)~p1$fBeach)
